Nuovi farmaci con DrugAI
Il 2023 ha visto un'impennata di interesse per i sistemi di IA generativa. Ma oltre a creare collage e ad assistere nella scrittura di e-mail, l'IA generativa potrebbe essere utilizzata anche per sviluppare nuovi farmaci.
L’intelligenza artificiale per creare nuove molecole
Nel 2023 le piattaforme di intelligenza artificiale generativa, da ChatGPT a Midjourney, hanno conquistato le prime pagine dei giornali. Ma l'intelligenza artificiale generativa può fare molto di più che creare collage di immagini e aiutare a scrivere e-mail: può anche progettare nuovi farmaci per il trattamento delle malattie.
Oggi gli scienziati utilizzano tecnologie avanzate per progettare nuovi composti farmaceutici sintetici con le giuste proprietà e caratteristiche. Tuttavia, i metodi attuali possono richiedere molto lavoro, tempo e costi.
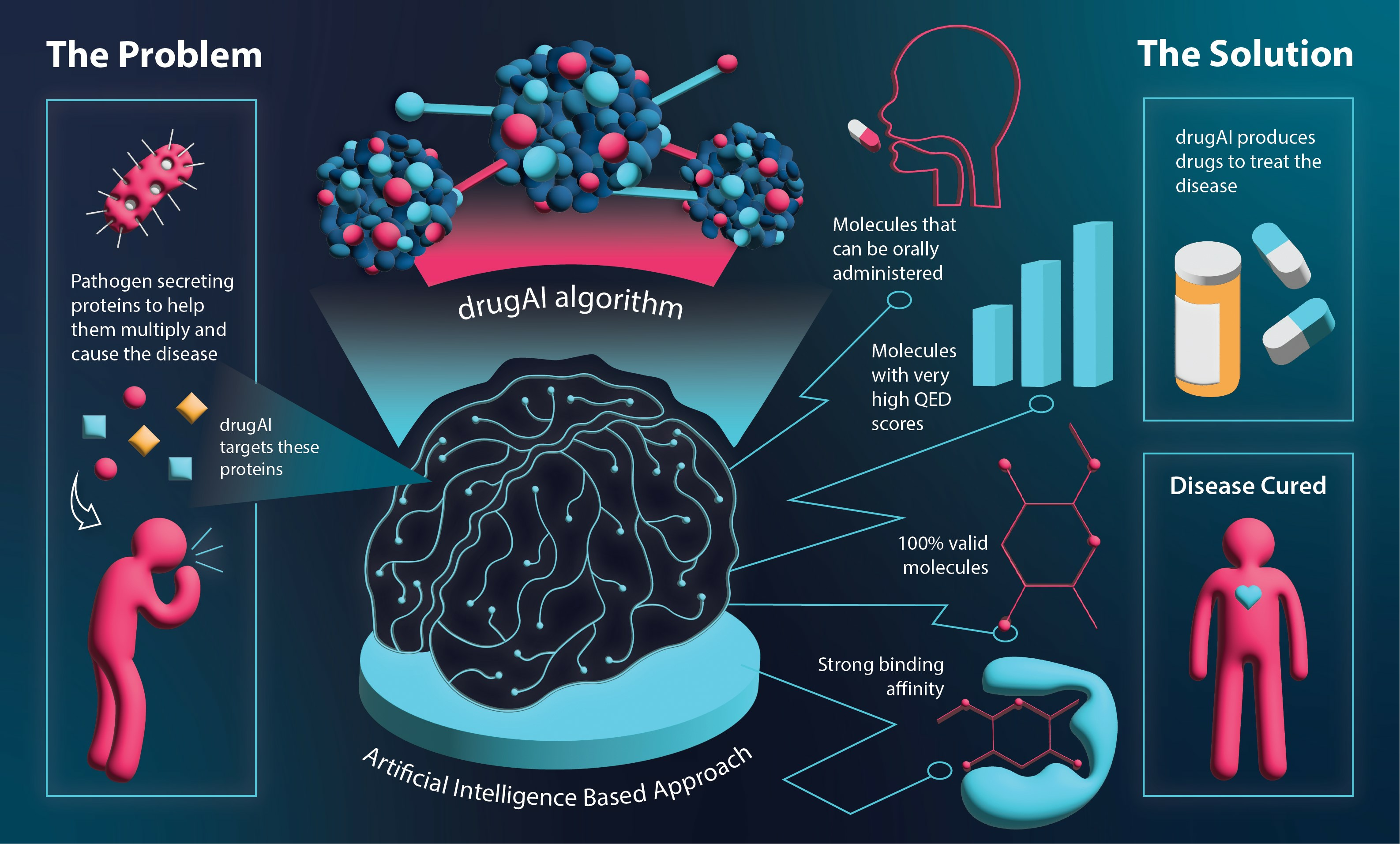
Ispirati dalla popolarità di ChatGPT e chiedendosi se questo approccio potesse accelerare il processo di progettazione dei farmaci, gli scienziati dello Schmid College of Science and Technology della Chapman University di Orange, in California, hanno deciso di creare il proprio modello di intelligenza artificiale. Dony Ang, Cyril Rakovski e Hagop Atamian hanno codificato un modello per apprendere una serie enorme di sostanze chimiche conosciute, il modo in cui si legano alle proteine bersaglio e le regole e la sintassi della struttura chimica e delle proprietà note.
Graphical abstract (photocredit: MDPI)
Il risultato finale è in grado di generare innumerevoli strutture molecolari uniche che seguono schemi chimici e biologici essenziali e si legano efficacemente ai loro bersagli - promettendo di accelerare enormemente il processo di identificazione di candidati farmaci validi per una vasta gamma di malattie, ad un costo inferiore di quello attuale.
DrugAI
Per creare questo modello innovativo, i ricercatori hanno integrato per la prima volta nel campo della bioinformatica e della chemoinformatica due tecniche di intelligenza artificiale all'avanguardia: la ben nota architettura "Encoder-Decoder Transformer" e il "Reinforcement Learning via Monte Carlo Tree Search" (RL-MCTS). La piattaforma, opportunamente denominata "DrugAI", consente agli utenti di inserire una sequenza proteica target (ad esempio, una proteina tipicamente coinvolta nella progressione del cancro). Il DrugAI, addestrato sui dati del database pubblico BindingDB, è in grado di generare strutture molecolari uniche partendo da zero e di affinare iterativamente i candidati, assicurando che i finalisti presentino una forte affinità di legame con i rispettivi bersagli farmacologici, fondamentale per l'efficacia dei potenziali farmaci. Il modello identifica 50-100 nuove molecole in grado di inibire queste particolari proteine.
I ricercatori hanno valutato le molecole generate da DrugAI in base a diversi criteri e hanno scoperto che i risultati di DrugAI erano di qualità simile a quelli ottenuti con altri due metodi comuni e, in alcuni casi, migliori. Hanno scoperto che i farmaci candidati di DrugAI avevano un tasso di validità del 100%, il che significa che nessuno dei farmaci generati era presente nel set di addestramento. I farmaci candidati di DrugAI sono stati anche misurati per la somiglianza con i farmaci, ovvero la somiglianza delle proprietà di un composto con quelle dei farmaci orali, e i farmaci candidati erano superiori di almeno il 42% e il 75% rispetto agli altri modelli. Inoltre, tutte le molecole generate da DrugAI hanno mostrato una forte affinità di legame con i rispettivi bersagli, paragonabile a quelle identificate attraverso i tradizionali approcci di screening virtuale.
Inoltre, gli scienziati hanno progettato l'algoritmo in modo da avere una struttura flessibile che permetta ai futuri ricercatori di aggiungere nuove funzioni.
Fonte: Ang D, Rakovski C, Atamian HS. De Novo Drug Design Using Transformer-Based Machine Translation and Reinforcement Learning of an Adaptive Monte Carlo Tree Search. Pharmaceuticals. 2024; 17(2):161. https://doi.org/10.3390/ph17020161