Pandemia influenzale del 1918 e COVID-19
Campioni polmonari conservati in archivi medici hanno permesso di produrre tre genomi del virus dell'influenza A che ha causato la pandemia globale del 1918.
La pandemia del 1918
L'ultima volta che un misterioso virus respiratorio ha messo in ginocchio il mondo è stato nel 1918, quando una pandemia, causata da un virus dell'influenza A H1N1, si è diffusa in tutto il mondo.
L'influenza spagnola, conosciuta anche come la spagnola o la grande influenza, fu una pandemia influenzale di natura virale e insolitamente mortale, che fra il 1918 e il 1920 uccise dalle decine al centinaio di milioni di persone nel mondo. Un fatto inconsueto fu che l'epidemia si diffuse in diverse ondate indipendenti dalle stagioni (per quanto riguarda l'emisfero settentrionale), mentre di solito l'influenza epidemica si diffonde in inverno. Anche se il periodo e il numero di ondate della Spagnola non sono coerenti a livello globale, c'è un certo consenso nel ritenere che la pandemia, negli USA e altre nazioni, abbia avuto tre onde distinte: la primavera del 1918, l'autunno del 1918 e l'inverno del 1918-1919. Alcuni autori considerano anche, in alcuni paesi, una quarta ondata del 1920-1921.
La seconda ondata della pandemia del 1918 fu molto più letale della prima. La prima ondata somigliava alle tipiche epidemie influenzali; i più a rischio furono coloro che erano anziani o già malati, mentre i più giovani e coloro che godevano di buona salute si riprendevano facilmente. Ma ad agosto, quando iniziò la seconda ondata in Francia, Sierra Leone e Stati Uniti, il virus era mutato in una forma molto più letale.
I passi evolutivi compiuti da quel virus H1N1 sono di particolare interesse oggi poiché la pandemia di COVID-19 continua.
I virus dell'influenza pandemica di 100 anni fa producono nuovi genomi
Per studiare l'evoluzione del virus, ricercatori hanno perlustrato gli archivi di musei medici europei alla ricerca di campioni “secolari” di polmoni contenenti RNA del virus del 1918. Gli scienziati hanno descritto le sequenze del genoma di tre virus H1N1 del 1918.
Grazie a nuove tecnologie, Patrono et al. hanno potuto sequenziare in modo efficiente un genoma completo e due genomi parziali di virus del 1918, trovati in campioni di polmone umano, conservati in formalina negli archivi del museo di Berlino. Due di questi genomi provenivano da persone note per essere morte nel giugno 1918, prima della seconda ondata della pandemia, consentendo agli autori di studiare i cambiamenti del virus nel tempo.
Il loro lavoro suggerisce che il virus dell'influenza del 1918 abbia acquisito mutazioni durante la prima ondata della pandemia nella primavera del 1918, nell'emisfero settentrionale, il che potrebbe spiegare perché la seconda ondata in autunno è stata così mortale.
Il riassortimento
Tutti i virus dell'influenza A umana, attualmente in circolazione, contengono geni che fanno risalire le loro origini al virus del 1918. Tuttavia, le origini di un gene che codifica per la proteina virale H1 (lo spike esterno preso di mira dalle risposte immunitarie dell'ospite), sono state meno chiare. Il genoma del virus dell'influenza A comprende otto segmenti che possono essere scambiati nella loro interezza quando due diversi ceppi del virus infettano una cellula contemporaneamente.
In questo processo, chiamato riassortimento, i geni dello stesso virus possono avere storie evolutive notevolmente diverse. Le differenze nella forma degli alberi filogenetici dedotti per diversi geni dell'influenza A, in precedenza hanno portato i ricercatori a supporre che si sia verificato un riassortimento tra il virus H1N1 del 1918 e un altro virus da cui ha avuto origine l'attuale proteina H1, creando un nuovo virus misto che ha generato successivi focolai stagionali. Tuttavia, Patrono et al. hanno osservato un tasso di evoluzione più rapido lungo un ramo dell'albero virale rispetto ad altre regioni. Questo lungo ramo rappresenta un divario di virus non campionati che circolarono tra il 1918 e i primi virus stagionali H1N1 sequenziati degli anni '30. Quando gli autori hanno tenuto conto della differenza di tasso, gli alberi per tutti i geni avevano schemi di ramificazione simili e hanno mostrato prove insufficienti di riassortimento. Questo ha suggerito invece che i virus dell'influenza stagionale attualmente in circolazione discendevano direttamente dai ceppi che avevano causato la pandemia del 1918.
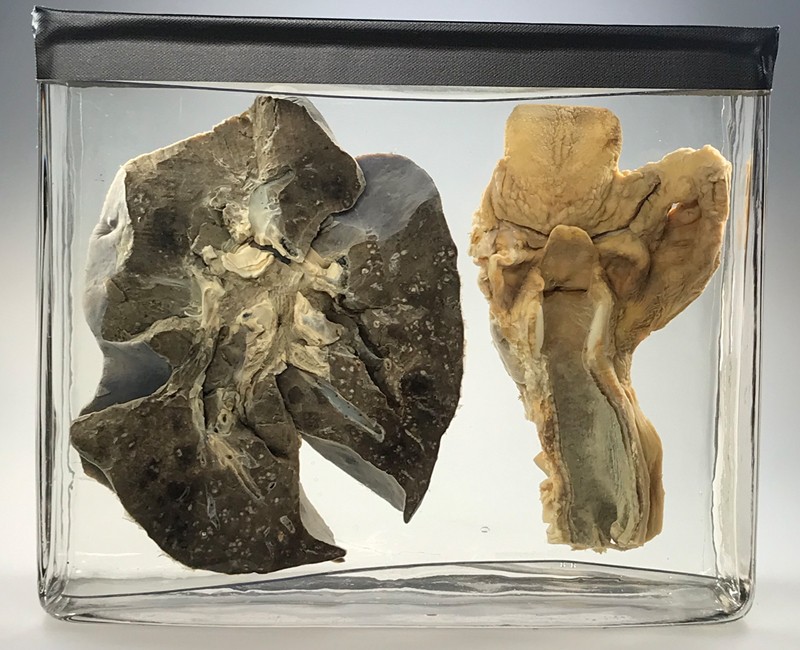
Polmoni secolari dagli archivi medici di Berlino.
Patrono et al. hanno utilizzato campioni di questi polmoni per generare sequenze genomiche per il virus dell'influenza A che ha causato la pandemia del 1918.
Crediti: Navena Widulin/Museo di storia medica della Charité di Berlino
Imparare dalle pandemie precedenti
Lezioni dalle pandemie precedenti, inclusa la loro progressione nelle ondate successive, hanno guidato le risposte politiche alle pandemie nel ventunesimo secolo. Il lavoro di Patrono e colleghi indica che i virus pandemici si evolvono dinamicamente durante le prime fasi, poiché ottimizzano la replicazione nelle persone. Molti paesi non hanno dato la priorità al sequenziamento genetico, durante le prime fasi della pandemia di COVID-19, perché si prevedeva che le mutazioni che riducono l'efficacia del vaccino emergessero solo dopo diversi anni. Se avessimo saputo di più sui primi mesi della pandemia del 1918, saremmo stati meno sorpresi dalle mutazioni del SARS-CoV-2 che hanno aumentato la replicazione virale e alterato il corso della pandemia durante il primo anno. Una sorveglianza genomica ad alto rendimento avrebbe dovuto essere stata stabilita molto prima negli Stati Uniti e in altri paesi.
Fonti: Patrono LV, Vrancken B, Budt M, Düx A, Lequime S, Boral S, Gilbert MTP, Gogarten JF, Hoffmann L, Horst D, Merkel K, Morens D, Prepoint B, Schlotterbeck J, Schuenemann VJ, Suchard MA, Taubenberger JK, Tenkhoff L, Urban C, Widulin N, Winter E, Worobey M, Schnalke T, Wolff T, Lemey P, Calvignac-Spencer S. Archival influenza virus genomes from Europe reveal genomic variability during the 1918 pandemic. Nat Commun. 2022 May 10;13(1):2314. doi: 10.1038/s41467-022-29614-9. PMID: 35538057; PMCID: PMC9090925.
Nelson MI, Ghedin E. 100-year-old pandemic flu viruses yield new genomes. Nature. 2022 Jul;607(7918):244-245. doi: 10.1038/d41586-022-01741-9. PMID: 35794385.
Wikipedia https://it.wikipedia.org/wiki/Influenza_spagnola